
Mục lục:
- Bước 1: Thiết lập Nhóm bộ nhớ đám mây của Google
- Bước 2: Định dạng dữ liệu của bạn và tạo tập dữ liệu Csv
- Bước 3: Tải ảnh quang phổ của bạn lên nhóm của bạn
- Bước 4: Tải lên Csv tập dữ liệu của bạn
- Bước 5: Tạo tập dữ liệu
- Bước 6: Tạo mô hình AutoML của bạn
- Bước 7: Kiểm tra mô hình của bạn
- Bước 8: Cài đặt mô hình của bạn vào ThinkBioT
- Tác giả John Day day@howwhatproduce.com.
- Public 2024-01-30 13:32.
- Sửa đổi lần cuối 2025-01-23 15:15.

ThinkBioT được thiết kế để "Cắm và chạy", với các Mô hình TensorFlow Lite tương thích với Edge TPU.
Trong tài liệu này, chúng tôi sẽ đề cập đến việc tạo quang phổ, định dạng dữ liệu của bạn và sử dụng Google AutoML.
Mã trong hướng dẫn này sẽ được viết bằng bash nên sẽ tương thích với nhiều nền tảng.
Sự phụ thuộc
- Tuy nhiên trước khi bắt đầu, bạn sẽ cần cài đặt Sox một chương trình âm thanh dòng lệnh tương thích với các thiết bị Windows, Mac và Linux.
- Nếu bạn đang sử dụng thiết bị Windows, cách dễ nhất để chạy các tập lệnh bash là thông qua Git, vì vậy tôi khuyên bạn nên tải xuống và cài đặt vì nó hữu ích theo nhiều cách,
- Để chỉnh sửa mã, hãy sử dụng trình soạn thảo yêu thích của bạn hoặc cài đặt NotePad ++ cho windows hoặc Atom cho các hệ điều hành khác.
** Nếu bạn có một mô hình TensorFlow hiện có hoặc muốn thử học chuyển tiếp với một mô hình hiện có, vui lòng tham khảo Tài liệu Google Coral.
Bước 1: Thiết lập Nhóm bộ nhớ đám mây của Google
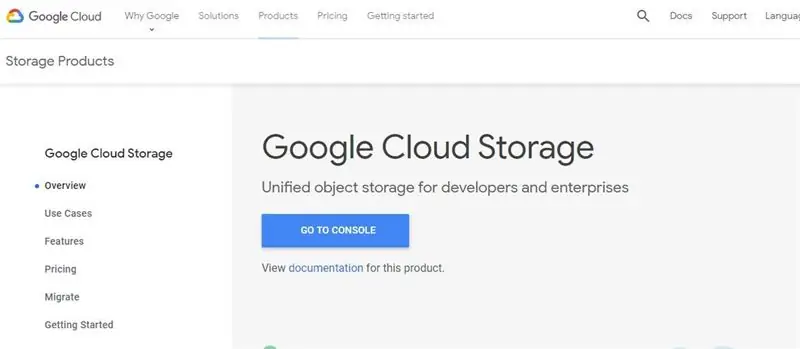
1. Đăng nhập vào tài khoản gmail của bạn (hoặc tạo một tài khoản nếu bạn chưa có tài khoản Google)
2. Đi tới trang bộ chọn dự án và tạo một dự án mới cho bạn các tệp mô hình và biểu đồ quang phổ. Bạn sẽ cần kích hoạt tính năng thanh toán để tiến bộ hơn nữa.
3. Truy cập https://cloud.google.com/storage/ và nhấn nút tạo nhóm ở đầu trang.
4. Nhập tên nhóm mong muốn của bạn và tạo nhóm chấp nhận cài đặt mặc định.
Bước 2: Định dạng dữ liệu của bạn và tạo tập dữ liệu Csv
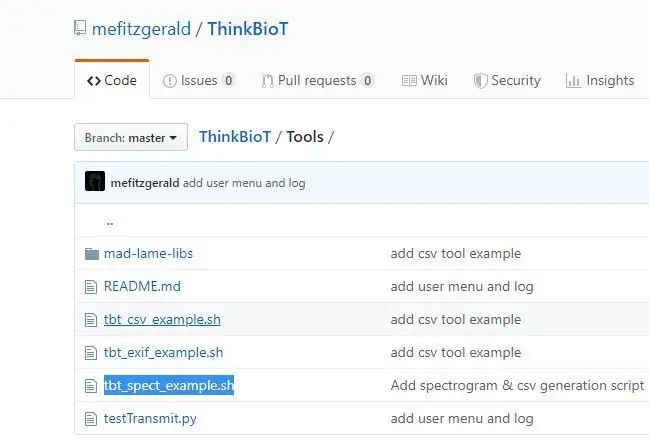
Tôi đã thiết kế một tập lệnh hữu ích để tạo tệp dataset.csv cần thiết để tạo mô hình của bạn. Tệp tập dữ liệu liên kết các hình ảnh trong nhóm của bạn với nhãn của chúng trong tập dữ liệu..
1. Tải xuống kho lưu trữ ThinkBioT từ GitHub và
2. Sao chép tệp tbt_spect_example.sh từ thư mục Công cụ vào một thư mục mới trên màn hình của bạn.
3. Thêm các tệp âm thanh bạn muốn sử dụng trong mô hình của mình, đặt chúng vào các thư mục có nhãn của chúng (nghĩa là bạn muốn chúng được sắp xếp vào. Ví dụ: nếu bạn muốn xác định chó hoặc mèo, bạn có thể có một thư mục con chó, với âm thanh sủa HOẶC thư mục có tên mèo với âm thanh mèo vv.
4. Mở tbt_spect_example.sh bằng Notepad ++ và thay thế "yourbucknamename" ở dòng 54 bằng tên Nhóm Google Storage của bạn. Ví dụ: nếu nhóm của bạn được gọi là myModelBucket, dòng sẽ được thay đổi thành
bucket = "gs: // myModelBucket / spectro-data /"
5. Chạy mã bằng cách nhập mã sau vào thiết bị đầu cuối Bash của bạn, mã sẽ chạy và tạo tệp csv nhãn của bạn và một thư mục được gọi là dữ liệu quang phổ trên mặt bàn của bạn với các biểu đồ quang phổ kết quả.
sh tbt_spect_example.sh
Bước 3: Tải ảnh quang phổ của bạn lên nhóm của bạn
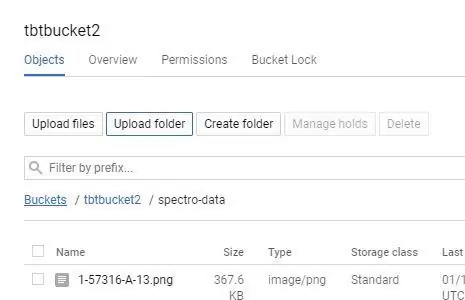
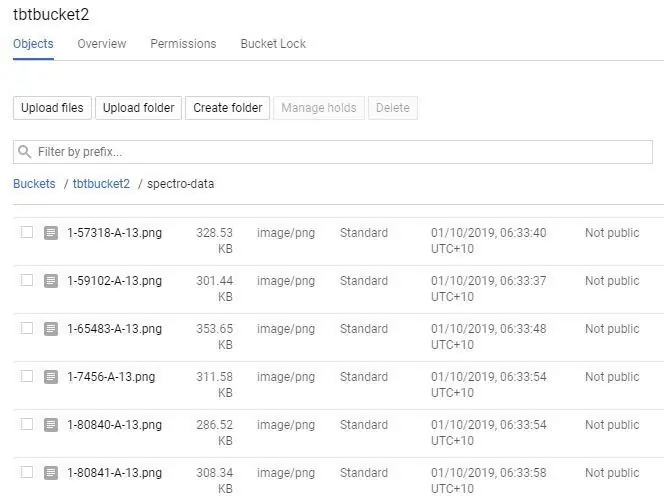
Có một số cách để tải lên Google Storage, đơn giản nhất là tải thư mục trực tiếp lên;
1. Nhấp vào tên nhóm của bạn trong trang Google Storage của bạn.
2. Chọn nút "TẢI THƯ MỤC" và chọn thư mục "spectro-data /" của bạn đã tạo ở bước cuối cùng.
HOẶC
2. Nếu bạn có một lượng lớn tệp, bạn có thể tạo thư mục "spectro-data /" theo cách thủ công bằng cách chọn "TẠO THƯ MỤC", sau đó điều hướng vào thư mục và chọn "TẢI LÊN FILES". Đây có thể là một lựa chọn tuyệt vời cho các tập dữ liệu lớn vì bạn có thể tải lên các biểu đồ quang phổ theo từng phần, thậm chí sử dụng nhiều máy tính để tăng tốc độ tải lên.
HOẶC
2. Nếu bạn là người dùng nâng cao, bạn cũng có thể tải lên qua Google Cloud Shell;
gsutil cp spectro-data / * gs: // your-bucket-name / spectro-data /
Bây giờ bạn sẽ có một thùng đầy các quang phổ khá đẹp!
Bước 4: Tải lên Csv tập dữ liệu của bạn
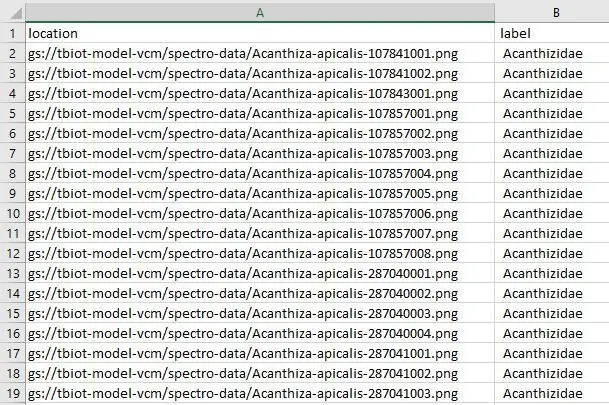
Bây giờ chúng tôi cần tải tệp model-label.csv lên thư mục "spectro-data /" của bạn trong Google Storage, về cơ bản nó giống như bước cuối cùng, bạn chỉ tải lên một tệp duy nhất thay vì nhiều tệp.
1. Nhấp vào tên nhóm của bạn trong trang Google Storage của bạn.
2. Chọn nút TẢI TỆP TỆP và chọn tệp model-label.csv mà bạn đã tạo trước đó.
Bước 5: Tạo tập dữ liệu
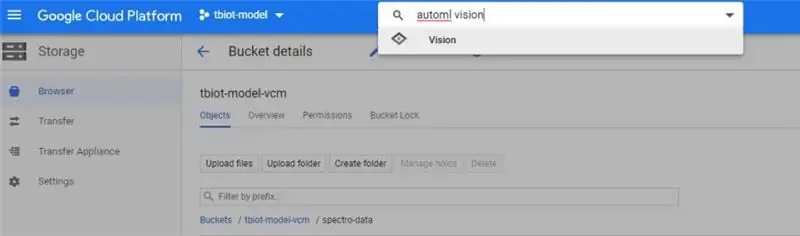
1. Đầu tiên, bạn sẽ cần tìm API AutoML VIsion, nó có thể hơi phức tạp! Cách dễ nhất là tìm kiếm "automl vision" trong thanh tìm kiếm của bộ lưu trữ Google Cloud của bạn (trong hình).
2. Khi bạn nhấp vào liên kết API, bạn sẽ cần kích hoạt API.
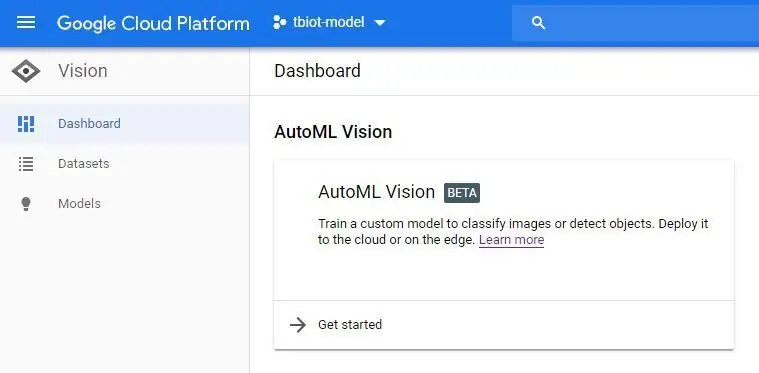
3. Bây giờ bạn sẽ ở trong Bảng điều khiển Tầm nhìn AutoML (trong hình), nhấp vào nút bộ dữ liệu mới và chọn Nhãn đơn và tùy chọn 'Chọn tệp CSV'. Sau đó, bạn sẽ bao gồm liên kết đến tệp model-label.csv trong nhóm lưu trữ của mình. Nếu bạn đã làm theo hướng dẫn này, nó sẽ như dưới đây
gs: //yourBucketName/spectro-data/model-labelsBal.csv
4. Sau đó nhấn tiếp tục để tạo tập dữ liệu của bạn. Có thể mất một thời gian để tạo.
Bước 6: Tạo mô hình AutoML của bạn
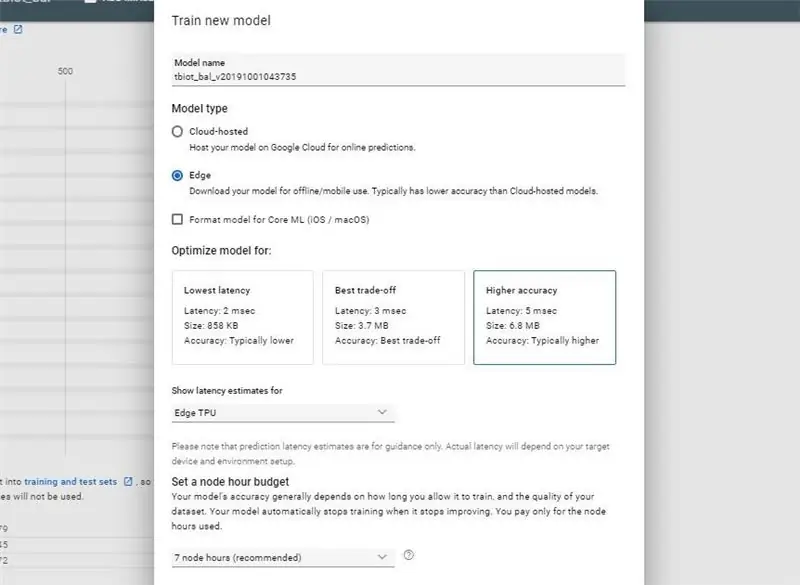

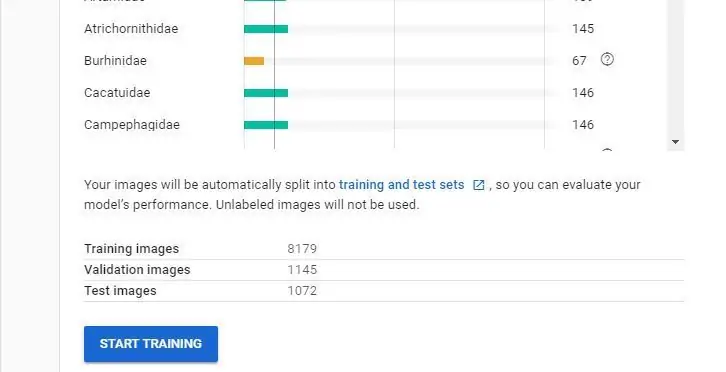
Sau khi bạn nhận được email thông báo cho bạn biết tập dữ liệu của bạn đã được tạo, bạn đã sẵn sàng tạo mô hình mới của mình.
- Nhấn nút TRAIN
- Chọn loại mô hình: Ước tính độ trễ của Edge và Model: Edge TPU và để các tùy chọn khác làm mặc định ban đầu, khó mà bạn có thể muốn thử nghiệm sau đó.
- Bây giờ mô hình của bạn sẽ được đào tạo, sẽ mất một khoảng thời gian và bạn sẽ nhận được email khi nó sẵn sàng để tải xuống.
Lưu ý: Nếu nút huấn luyện của bạn không khả dụng, bạn có thể gặp sự cố với tập dữ liệu của mình. Nếu bạn có ít hơn 10 trong mỗi lớp (nhãn), hệ thống sẽ không cho phép bạn Huấn luyện Mô hình, vì vậy bạn có thể phải thêm hình ảnh bổ sung. Bạn nên xem qua Google AutoML Video nếu bạn cần làm rõ.
Bước 7: Kiểm tra mô hình của bạn
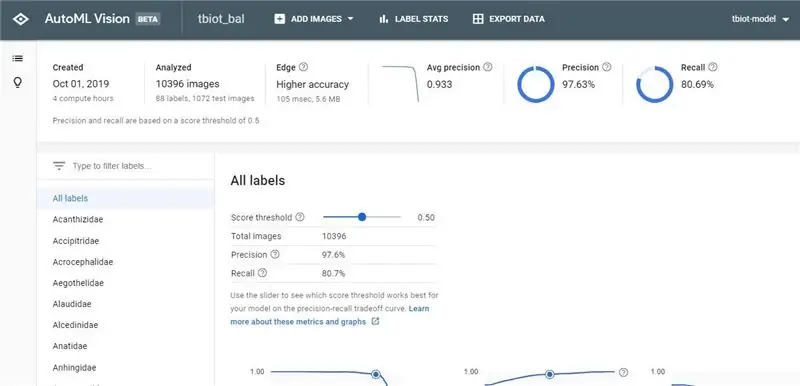
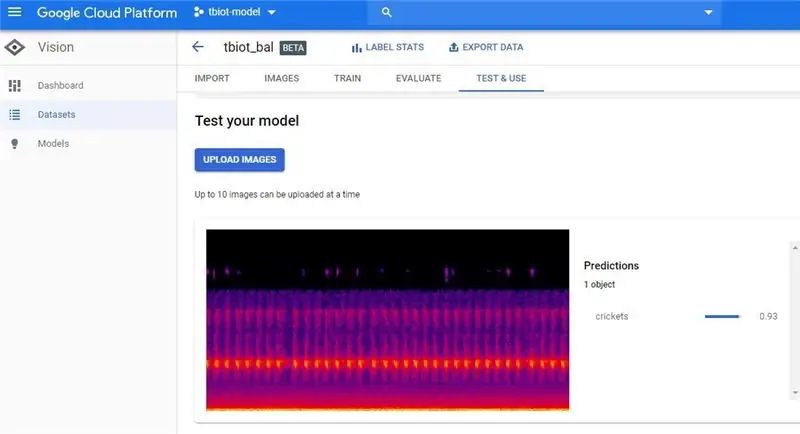
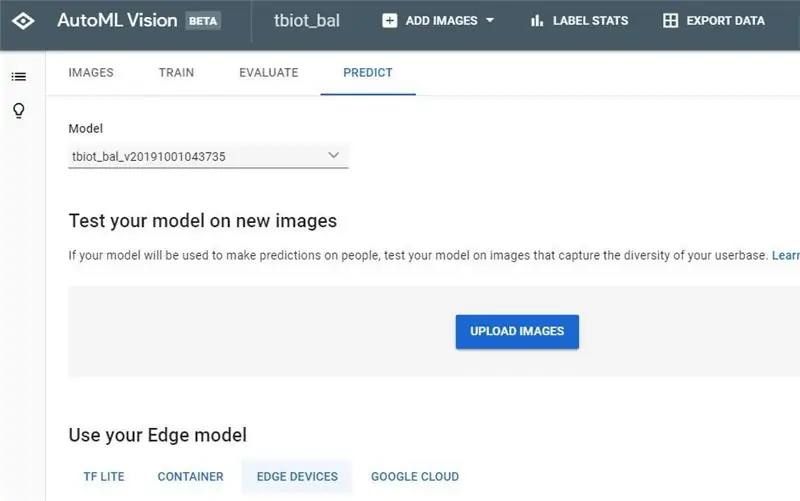
Sau khi bạn nhận được email hoàn thành mô hình của mình, hãy nhấp vào liên kết để quay lại API AutoML Vision.
1. Bây giờ bạn sẽ có thể xem kết quả của mình và ma trận nhầm lẫn cho mô hình của bạn.
2. Bước tiếp theo là kiểm tra Model của bạn, chuyển đến 'TEST & USE' hoặc 'PREDICT', kỳ lạ là dường như có 2 GUI của người dùng, cả hai đều như tôi đã hình dung, nhưng các tùy chọn đều có chức năng giống nhau.
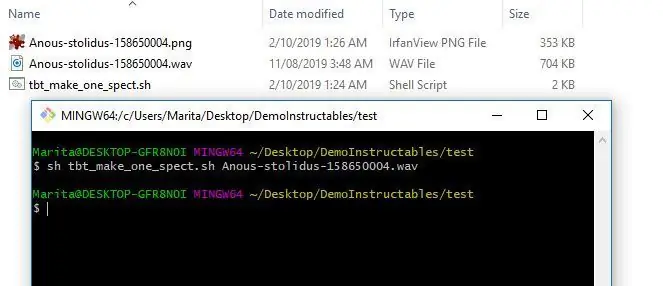
3. Bây giờ bạn có thể tải lên một quang phổ thử nghiệm. Để tạo một biểu đồ quang phổ duy nhất, bạn có thể sử dụng chương trình tbt_make_one_spect.sh từ ThinkBioT Github. Chỉ cần thả nó vào một thư mục có wav mà bạn muốn chuyển đổi thành một biểu đồ quang phổ, mở cửa sổ Git Bash (hoặc thiết bị đầu cuối) và sử dụng mã bên dưới, thay thế tên tệp của bạn.
sh tbt_make_one_spect.sh yourWavName.wav
4. Bây giờ chỉ cần tải lên quang phổ và kiểm tra kết quả của bạn!
Bước 8: Cài đặt mô hình của bạn vào ThinkBioT
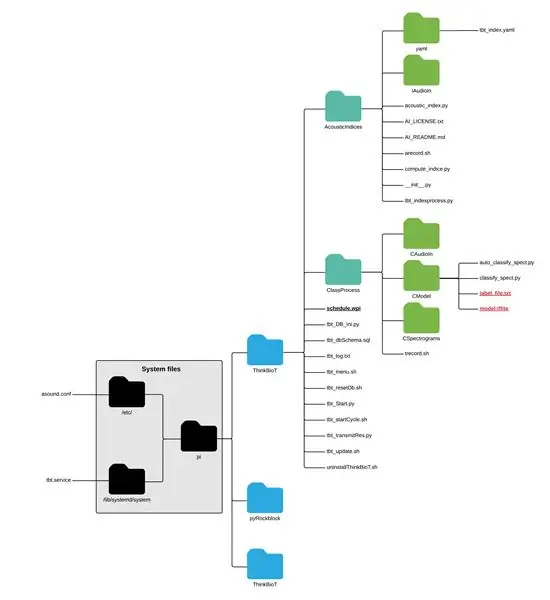
Để sử dụng mô hình sáng bóng mới của bạn, chỉ cần thả mô hình và tệp txt vào thư mục CModel;
pi> ThinkBioT> ClassProcess> CModel
Bây giờ bạn đã sẵn sàng để sử dụng ThinkBioT:)
** NB ** Nếu bạn đang sử dụng mô hình của mình bên ngoài khuôn khổ ThinkBioT, bạn sẽ cần chỉnh sửa tài liệu nhãn của mình bằng cách thêm âm sắc vào đầu mỗi dòng vì các trình thông dịch tflite mới nhất được tích hợp sẵn chức năng "nhãn đọc" giả định rằng chúng ở đó. Tôi đã viết một hàm tùy chỉnh trong khung ThinkBioT Class_spect.py như một công việc mà bạn được hoan nghênh sử dụng trong mã của riêng mình:)
def ReadLabelFile (file_path):
counter = 0 với open (file_path, 'r', encoding = 'utf-8') là f: lines = f.readlines () ret = {} cho dòng trong dòng: ret [int (counter)] = line.strip () counter = counter + 1 return ret
Đề xuất:
Esp8266 dựa trên Boost Converter với giao diện người dùng Blynk tuyệt vời với bộ điều chỉnh phản hồi: 6 bước
Esp8266 dựa trên Boost Converter với giao diện người dùng Blynk tuyệt vời với bộ điều chỉnh phản hồi: Trong dự án này, tôi sẽ chỉ cho bạn một cách hiệu quả và phổ biến cách tăng điện áp DC. Tôi sẽ cho bạn thấy việc xây dựng một bộ chuyển đổi tăng cường có thể dễ dàng như thế nào với sự trợ giúp của Nodemcu. Hãy xây dựng nó. Nó cũng bao gồm một vôn kế trên màn hình và một phản hồi
Arduino Bắt đầu với Phần cứng và Phần mềm & Hướng dẫn Arduino: 11 bước

Arduino Bắt đầu với Phần cứng và Phần mềm & Hướng dẫn về Arduino: Ngày nay, các Nhà sản xuất, Nhà phát triển đang ưa thích Arduino để phát triển nhanh chóng việc tạo mẫu của các dự án.Arduino là một nền tảng điện tử mã nguồn mở dựa trên phần cứng và phần mềm dễ sử dụng. Arduino có cộng đồng người dùng rất tốt. Bảng Arduino d
Phần 1. Cấu tạo phần cứng cảm biến âm thanh sinh học tự trị ThinkBioT: 13 bước

Phần 1. Phần cứng cảm biến âm thanh sinh học tự trị ThinkBioT Xây dựng: ThinkBioT nhằm mục đích cung cấp khung phần mềm và phần cứng, được thiết kế như một xương sống công nghệ để hỗ trợ nghiên cứu sâu hơn, bằng cách xử lý các tác vụ thu thập dữ liệu, tiền xử lý, truyền dữ liệu và trực quan hóa cho phép nhà nghiên cứu
Đồng hồ kỹ thuật số & nhị phân trong 8 chữ số X 7 phân đoạn Màn hình LED: 4 bước (có hình ảnh)

Đồng hồ kỹ thuật số & nhị phân ở 8 chữ số X 7 phân đoạn Màn hình LED: Đây là phiên bản nâng cấp của tôi về Đồng hồ kỹ thuật số & Đồng hồ nhị phân sử dụng Màn hình LED 8 chữ số x 7 đoạn. Tôi muốn cung cấp các tính năng mới cho các thiết bị thông thường, đồng hồ đặc biệt và trong trường hợp này, việc sử dụng màn hình 7 Seg cho Đồng hồ nhị phân là không bình thường và nó
Chụp ảnh toàn cảnh với phần mềm miễn phí và phần cứng giá rẻ: 6 bước

Chụp ảnh toàn cảnh với phần mềm miễn phí và phần cứng giá rẻ: Ảnh toàn cảnh được sử dụng để tạo ra hình ảnh của những cảnh quá lớn để vừa với ống kính máy ảnh thông thường hoặc thậm chí quá lớn để mắt người có thể nhìn thấy cùng một lúc. Hầu hết các ảnh toàn cảnh nổi tiếng là ảnh chụp phong cảnh ngoài trời về các đặc điểm địa chất hoặc bầu trời thành phố
