Mục lục:
- Tác giả John Day day@howwhatproduce.com.
- Public 2024-01-30 13:30.
- Sửa đổi lần cuối 2025-01-23 15:14.
Bởi: Phúc Lâm, Paul Yeung, Eric Reyes
Thừa nhận rằng các sai sót trong phân đoạn phổi sẽ tạo ra thông tin sai lệch liên quan đến việc xác định vùng bệnh và có thể ảnh hưởng trực tiếp đến quá trình chẩn đoán. Các kỹ thuật hỗ trợ của máy tính hiện đại không mang lại kết quả chính xác khi các bệnh phổi có hình dạng khó khăn. Những hình dạng bất thường này có thể do tràn dịch màng phổi, hợp nhất, v.v. Áp dụng kỹ thuật phân đoạn phổi, trong đó ranh giới của phổi được tách biệt khỏi mô lồng ngực xung quanh, ứng dụng của chúng tôi có thể xác định ranh giới với ngưỡng đầu vào của người dùng để cung cấp các chế độ xem hoàn toàn có thể tùy chỉnh hình dạng của phổi, Mục đích của dự án MatLab này là tạo ra một ứng dụng phân đoạn phổi tương tác thân thiện với người dùng để phát hiện các tình trạng bệnh lý của hình ảnh X-quang của phổi. Mục đích của chúng tôi là tạo ra một cách hiệu quả hơn để minh họa và xác định các phổi bất thường nhằm cung cấp cho các bác sĩ và bác sĩ X quang một cách đáng tin cậy hơn để chẩn đoán các bệnh về phổi. Bằng cách sử dụng công cụ thiết kế ứng dụng trong MatLab, chương trình được thiết kế để hoạt động đặc biệt với chụp X-quang ngực và chụp cắt lớp vi tính (CT), nhưng nó cũng được thử nghiệm để hoạt động với quét MRI.
Các hướng dẫn bên dưới bao gồm kỹ thuật lọc nhiễu của chúng tôi (bộ lọc Wiener thông thấp) cũng như ngưỡng hình ảnh (bằng cách sử dụng biểu đồ cường độ của hình ảnh thang độ xám) và sử dụng gradient hình thái học (sự khác biệt giữa độ giãn nở và độ xói mòn của hình ảnh) để xác định một khu vực quan tâm. Sau đó, hướng dẫn sẽ giải thích cách chúng tôi tích hợp tất cả các yếu tố vào giao diện người dùng đồ họa (GUI).
Ghi chú:
1). Dự án này được lấy cảm hứng từ một bài báo nghiên cứu: "Phân tích và phân tích hình ảnh của phổi bất thường ở CT: Phương pháp tiếp cận hiện tại, thách thức và xu hướng tương lai". Có thể tìm thấy ở đây
2). Chúng tôi đang sử dụng hình ảnh tia X từ NIH: Clinical Center. Liên kết có thể được tìm thấy ở đây
3). Bạn có thể tìm thấy trợ giúp của nhà thiết kế ứng dụng tại đây
4). Trước khi chạy mã: bạn cần thay đổi đường dẫn Dir (ở dòng 34) thành thư mục tệp của bạn và loại hình ảnh (dòng 35) (chúng tôi đang phân tích *.png).
Bước 1: Bước 1: Tải hình ảnh
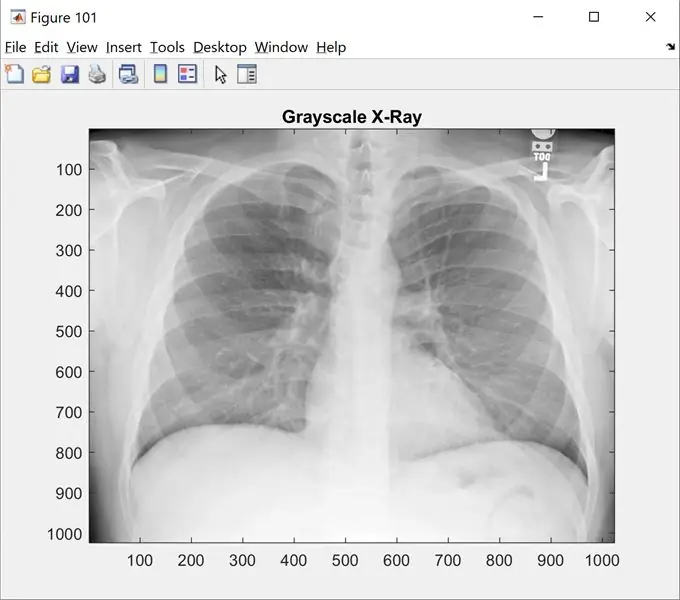
Bước này sẽ hiển thị cho bạn hình ảnh gốc ở thang màu xám. Thay đổi 'name_of_picture.png' thành tên hình ảnh của bạn
sạch; clc; đóng tất cả;
%% Đang tải hình ảnh
raw_x_ray = 'name_of_picture.png';
I = imread (raw_x_ray);
hình (101);
imshow (tôi);
bản đồ màu (xám);
title ('X-Ray thang độ xám');
Bước 2: Bước 2: Lọc tiếng ồn và biểu đồ
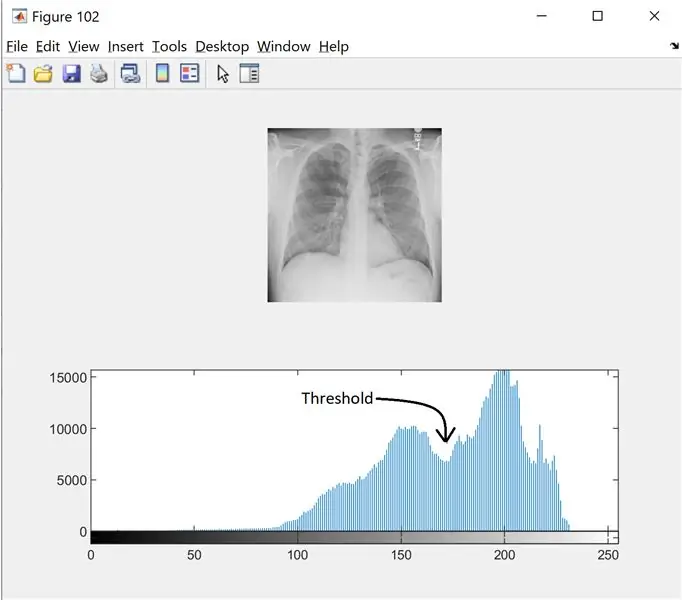
Để tìm ngưỡng cho hình ảnh thang màu xám, chúng tôi nhìn vào biểu đồ để xem có các chế độ riêng biệt hay không. Đọc thêm tại đây
I = wiener2 (I, [5 5]);
hình (102);
subplot (2, 1, 1);
imshow (tôi);
subplot (2, 1, 2);
imhist (I, 256);
Bước 3: Bước 3: Đặt ngưỡng
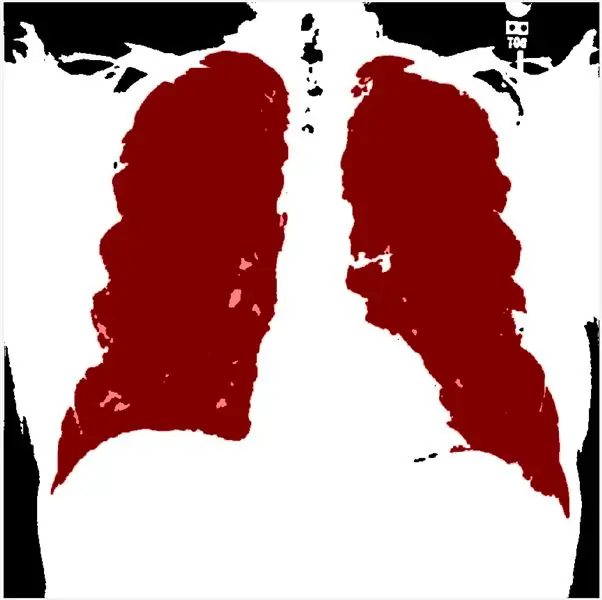
Bước này cho phép bạn đặt ngưỡng theo biểu đồ. morphologicalGradient sẽ làm nổi bật vùng quan tâm bằng màu đỏ và các đường ranh giới chức năng phủ lên hình ảnh được phác thảo và lọc của phổi bằng màu đỏ.
Bằng cách sử dụng các regionprops, chúng ta có thể chính xác hóa các mảng solidility và sắp xếp chúng theo thứ tự giảm dần. Tiếp theo, tôi mã hóa hình ảnh sclae màu xám và áp dụng phương pháp gradient hình thái học và mLoren Shurasking để làm nổi bật vùng quan tâm (ROI). Bước tiếp theo là đảo ngược hình ảnh để ROI của phổi có màu trắng trên nền đen. Tôi sử dụng hàm showMaskAsOverlay để hiển thị 2 mặt nạ. Lưu ý: mã được lấy cảm hứng từ Loren Shure, liên kết.
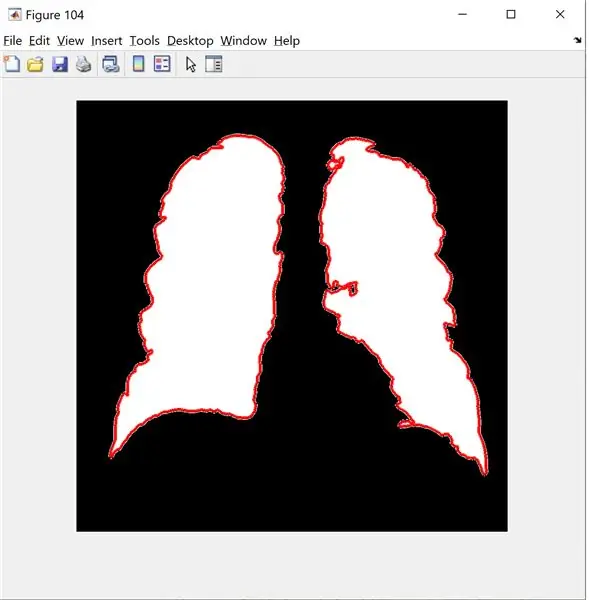
Cuối cùng, tôi tạo đường viền màu đỏ bằng cách sử dụng bwbwboundaries và che hình ảnh bộ lọc và các ranh giới.
a_thresh = I> = 172; % đặt ngưỡng này
[labelImage, numberOfBlobs] = bwlabel (a_thresh);
props = regionprops (a_thresh, 'all');
sortedSolidity = sort ([props. Solidity], 'con');
SB = sortedSolidity (1);
nếu SB == 1% SB chỉ chấp nhận độ rắn == 1 lọc bỏ xương
binaryImage = coeinarize (I); hình (103);
imshow (binaryImage); bản đồ màu (xám);
SE = strel ('hình vuông', 3);
morphologicalGradient = imsubtract (imdilate (binaryImage, SE), imerode (binaryImage, SE));
mặt nạ = chống thấm (morphologicalGradient, 0,03);
SE = strel ('hình vuông', 2);
mask = imclose (mặt nạ, SE);
mask = imfill (mặt nạ, 'lỗ');
mask = bwareafilt (mặt nạ, 2); % kiểm soát số khu vực hiển thị
notMask = ~ mặt nạ;
mask = mặt nạ | bwpropfilt (notMask, 'Area', [-Inf, 5000 - eps (5000)]);
showMaskAsOverlay (0,5, mặt nạ, 'r'); % bạn phải tải xuống ứng dụng / chức năng showMaskAsOverlay
BW2 = imfill (binaryImage, 'lỗ');
new_image = BW2;
new_image (~ mặt nạ) = 0; % đảo ngược nền và lỗ
B = bwboundaries (new_image); % chỉ có thể chấp nhận 2 thứ nguyên
hình (104);
imshow (hình ảnh mới);
Giữ lấy
đường biên (B);
kết thúc
Bước 4: Tạo GUI
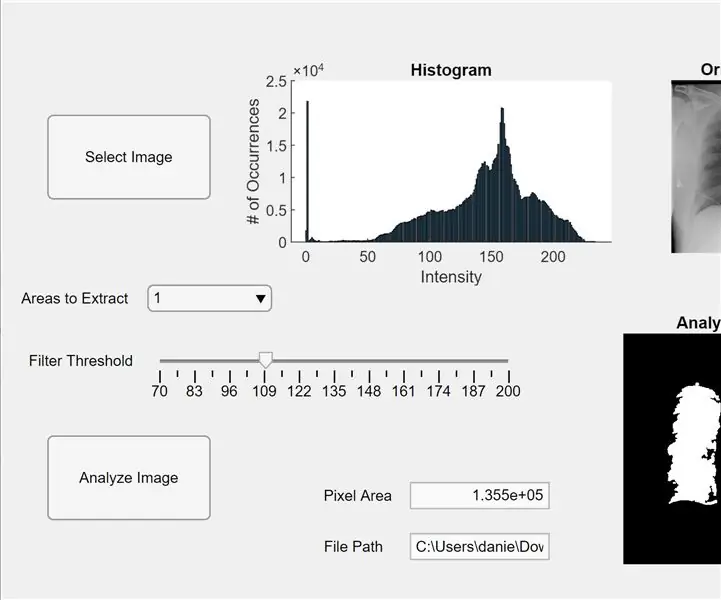
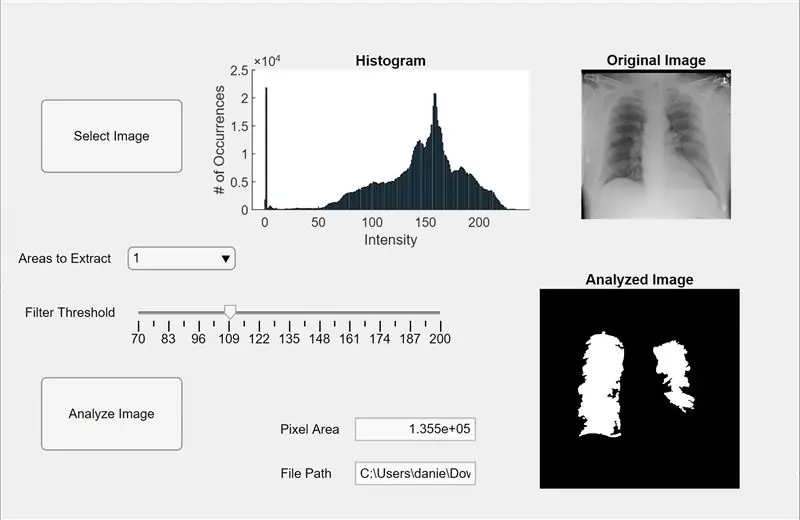
Bây giờ, chúng tôi tích hợp mã trước đó vào ứng dụng MATLAB. Mở Trình thiết kế ứng dụng trong MATLAB (Mới> Ứng dụng). Đầu tiên, chúng tôi thiết kế giao diện bằng cách nhấp-giữ và kéo theo ba trục vào không gian làm việc trung tâm. Tiếp theo, chúng ta nhấp-giữ-kéo hai nút, một trường chỉnh sửa (văn bản), một trường chỉnh sửa (số), một thanh trượt và một menu thả xuống. Hai trục, mỗi trục sẽ hiển thị bản xem trước và phân tích hình ảnh, và các trục thứ ba sẽ hiển thị biểu đồ pixel cho hình ảnh “đã chọn” xem trước. Hộp trường chỉnh sửa (văn bản) sẽ hiển thị đường dẫn tệp của hình ảnh đã chọn và trường chỉnh sửa (số) sẽ hiển thị vùng pixel được phát hiện của phổi.
Bây giờ chuyển từ chế độ xem thiết kế sang chế độ xem mã trong App Designer. Nhập mã mã cho các thuộc tính bằng cách nhấp vào nút “Thuộc tính” màu đỏ có dấu cộng bằng nó. Khởi tạo các thuộc tính I, ngưỡng và vùngToExtract như trong đoạn mã được cung cấp bên dưới. Tiếp theo, nhấp chuột phải vào một nút ở phía trên bên phải của không gian làm việc (Trình duyệt Thành phần) và đi từ Callbacks> Go to… callback. Thêm mã cho “chức năng SelectImageButtonPushing (ứng dụng, sự kiện).” Mã này cho phép bạn chọn một hình ảnh để phân tích từ máy tính của bạn bằng cách sử dụng uigetfile. Sau khi chọn hình ảnh, hình ảnh xem trước sẽ xuất hiện dưới các trục kèm theo biểu đồ. Sau đó, nhấp chuột phải vào nút khác và lặp lại quy trình tương tự để tạo hàm gọi lại.
Thêm mã bên dưới “function AnalyzeImageButtonPushing (ứng dụng, sự kiện).” Mã này sẽ thực hiện đếm pixel và phát hiện đốm màu trên hình ảnh xem trước khi nút phân tích hình ảnh (bất kỳ nút nào bạn nhấp chuột phải cho mã này). Sau khi lập trình các nút, bây giờ chúng ta sẽ lập trình thanh trượt và menu thả xuống. Nhấp chuột phải vào thanh trượt, tạo một hàm gọi lại và thêm mã bên dưới “function FilterThresholdSliderValueChanged (app, event)” cho đến khi kết thúc. Điều này cho phép thanh trượt điều chỉnh ngưỡng cường độ xám.
Tạo một hàm gọi lại cho menu thả xuống và thêm mã bên dưới “function AreastoExtractDropDownValueChanged (ứng dụng, sự kiện)” để cho phép menu thả xuống sửa đổi số lượng đốm màu hiển thị trên các trục hình ảnh được phân tích. Bây giờ, hãy nhấp vào từng thực thể trong Trình duyệt Thành phần và thay đổi các thuộc tính của chúng theo ý thích của bạn, chẳng hạn như thay đổi tên của các thực thể, loại bỏ các trục và thay đổi tỷ lệ. Kéo và thả các thực thể của Trình duyệt Thành phần trong Dạng xem Thiết kế vào một bố cục chức năng và dễ hiểu. Giờ đây, bạn có một ứng dụng trong MATLAB có thể phân tích hình ảnh của phổi cho diện tích pixel!
thuộc tính (Access = private) I = ; % tệp hình ảnh
ngưỡng = 257; ngưỡng% đối với cường độ xám hai cực
khu vựcToExtract = 2;
kết thúc
function SelectImageButtonPushing (ứng dụng, sự kiện)
clc; Dir = 'C: / Users / danie / Downloads / images_004 / images'; % xác định tệp bất biến "tiền tố"
[imageExt, path] = uigetfile ('*. png'); % lấy phần biến của tên hình ảnh
imageName = [Dir filesep imageExt]; % nối chuỗi bất biến và biến
app. I = imread (imageName); % đọc hình ảnh
imshow (app. I, 'parent', app. UIAxes); % hiển thị hình ảnh
app. FilePathEditField. Value = đường dẫn; % hiển thị đường dẫn tệp về nơi hình ảnh gốc đến từ
kết thúc
function AnalyzeImageButtonPushing (ứng dụng, sự kiện)
originalImage = ứng dụng. I;
originalImage = wiener2 (app. I, [5 5]); % dot-remove filter
biểu đồ (app. AxesHistogram, app. I, 256); % hiển thị biểu đồ của hình ảnh
a_thresh = originalImage> = app.threshold; % đặt ngưỡng này
labelImage = bwlabel (a_thresh);
props = regionprops (a_thresh, 'all');
sortedSolidity = sort ([props. Solidity], 'con');
SB = sortedSolidity (1);
nếu SB == 1% SB chỉ chấp nhận độ rắn == 1 lọc bỏ xương
SE = strel ('hình vuông', 3);
morphologicalGradient = imsubtract (imdilate (labelImage, SE), imerode (labelImage, SE));
mặt nạ = embinarize (morphologicalGradient, 0,03);
SE = strel ('hình vuông', 2);
mask = imclose (mặt nạ, SE);
mask = imfill (mặt nạ, 'lỗ');
mask = bwareafilt (mask, app.regionsToExtract);
% kiểm soát số khu vực hiển thị
notMask = ~ mặt nạ;
mask = mặt nạ | bwpropfilt (notMask, 'Area', [-Inf, 5000 - eps (5000)]);
BW2 = imfill (labelImage, 'lỗ');
new_image = BW2;
new_image (~ mặt nạ) = 0;
B = bwboundaries (new_image); % chỉ có thể chấp nhận 2 thứ nguyên imshow (new_image, 'parent', app. UIAxes2);
giữ (app. UIAxes2, 'trên');
đường biên (B);
set (gca, 'YDir', 'reverse');
lungArea = bwarea (new_image);
app. PixelAreaEditField. Value = lungArea;
kết thúc
kết thúc
function FilterThresholdSliderValueChanged (ứng dụng, sự kiện)
app.threshold = app. FilterThresholdSlider. Value;
kết thúc
function AreastoExtractDropDownValueChanged (ứng dụng, sự kiện) stringNumber = app. AreastoExtractDropDown. Value;
app.regionsToExtract = str2double (stringNumber);
kết thúc
kết thúc
Đề xuất:
Máy phân phối rượu dạng gel tự động với Esp32: 9 bước

Máy rút rượu gel tự động với Esp32: Trong hướng dẫn, chúng ta sẽ xem cách tạo một nguyên mẫu hoàn chỉnh, để lắp ráp máy phân phối rượu gel tự động với esp32, nó sẽ bao gồm lắp ráp từng bước, mạch điện tử và mã nguồn được giải thích tất cả từng bước bươc chân
Mạch phân phối nước rửa tay / DIY [Không tiếp xúc]: 10 bước
![Mạch phân phối nước rửa tay / DIY [Không tiếp xúc]: 10 bước Mạch phân phối nước rửa tay / DIY [Không tiếp xúc]: 10 bước](https://i.howwhatproduce.com/images/001/image-1630-j.webp)
Mạch phân phối nước rửa tay / DIY [Không liên hệ]: Bởi Hesam Moshiri, hesam.moshiri@gmail.com Tính năng Độ ổn định cao và không nhạy cảm với ánh sáng xung quanh Vỏ bọc acrylic (plexiglass) được cắt bằng laser Tiết kiệm chi phí Khả năng kiểm soát dòng chảy của hộp đựng nước rửa tay / alcohol (hiệu suất)
Bộ phân phối mặt nạ Arduino: 11 bước

Bộ phân phối mặt nạ Arduino: Đầu tiên, tôi biết điều này trông hơi kỳ lạ, nhưng vì lợi ích của chức năng, nó phải trông giống như một chiếc USS Enterprise màu trắng nhỏ. khử trùng khẩu trang của bạn trên p
Phân phối điện trên ô tô khe cắm kỹ thuật số: 9 bước (có hình ảnh)

Phân phối điện cho ô tô có khe cắm kỹ thuật số: Bạn đã bao giờ tự xây dựng cho mình một bố trí ô tô có khe cắm lớn và nhận thấy rằng những chiếc xe dường như không có cùng hiệu suất? Hay bạn ghét nó khi cuộc đua của bạn bị gián đoạn bởi những chiếc xe dừng lại vì khớp không tốt? Tài liệu hướng dẫn này sẽ chỉ cho bạn cách
Đồng hồ kỹ thuật số & nhị phân trong 8 chữ số X 7 phân đoạn Màn hình LED: 4 bước (có hình ảnh)

Đồng hồ kỹ thuật số & nhị phân ở 8 chữ số X 7 phân đoạn Màn hình LED: Đây là phiên bản nâng cấp của tôi về Đồng hồ kỹ thuật số & Đồng hồ nhị phân sử dụng Màn hình LED 8 chữ số x 7 đoạn. Tôi muốn cung cấp các tính năng mới cho các thiết bị thông thường, đồng hồ đặc biệt và trong trường hợp này, việc sử dụng màn hình 7 Seg cho Đồng hồ nhị phân là không bình thường và nó
