Mục lục:
- Bước 1: Tạo Ma trận cận kề
- Bước 2: Tạo mối quan hệ
- Bước 3: Thêm Thống kê Bệnh tật
- Bước 4: Chọn ngẫu nhiên Cơ hội Một Người đã Tiêm chủng và Chưa được Tiêm chủng Có thể Bị Nhiễm
- Bước 5: Tạo ma trận những người chưa được tiêm chủng và bị nhiễm bệnh từ thông tin ban đầu
- Bước 6: Vẽ đồ thị ban đầu
- Bước 7: Mô phỏng sự tiến triển của nhiễm trùng
- Bước 8: Sử dụng lý thuyết Monte Carlo
- Bước 9: Tạo tệp ('Nhiễm trùngSim.m') với Mô phỏng thành một chức năng
- Bước 10: Tính Tỷ lệ người chưa được tiêm chủng và người chưa được tiêm chủng bị nhiễm bệnh
- Bước 11: Tạo một biến đầu ra trong hàm 'Nhiễm trùngSim.m' của bạn
- Bước 12: Tạo Menu để Lấy Điều kiện ban đầu của Mô phỏng từ Người dùng
- Bước 13: Chọn% số người chưa được tiêm chủng và tính toán tỷ lệ trung bình của số người chưa được tiêm chủng và bị nhiễm bệnh cho phần trăm được chọn
- Bước 14: Biểu đồ: 'Xu hướng lây nhiễm ở những bệnh nhân chưa được tiêm chủng Vs. Được chủng ngừa cho các bệnh được chỉ định '
- Bước 15: Sản phẩm cuối cùng: Mô phỏng trông như thế nào
- Tác giả John Day day@howwhatproduce.com.
- Public 2024-01-30 13:35.
- Sửa đổi lần cuối 2025-01-23 15:15.
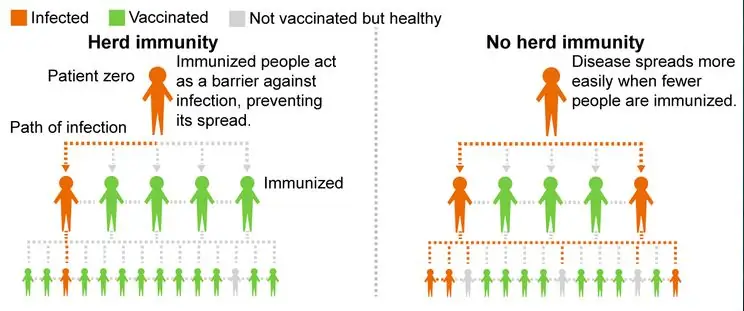
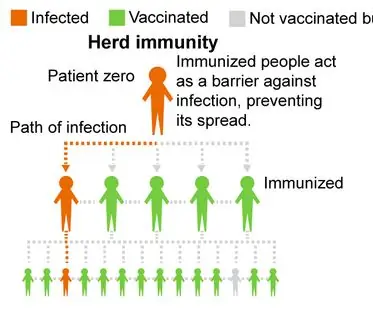
Tổng quan về dự án:
Dự án của chúng tôi khám phá khả năng miễn dịch của đàn và hy vọng sẽ khuyến khích mọi người tiêm phòng để giảm tỷ lệ nhiễm bệnh trong cộng đồng của chúng tôi. Chương trình của chúng tôi mô phỏng cách một căn bệnh lây nhiễm sang một quần thể với tỷ lệ phần trăm khác nhau giữa tỷ lệ được tiêm chủng và chưa được tiêm chủng. Nó cho thấy khả năng miễn dịch của đàn bằng cách cho thấy số lượng tăng lên của quần thể được tiêm chủng có thể làm giảm số người bị ảnh hưởng như thế nào.
Chúng tôi mô hình hóa điều này trong Matlab bằng cách sử dụng các khái niệm lý thuyết đồ thị. Lý thuyết đồ thị là một cách toán học để biểu diễn các mối quan hệ giữa các đối tượng. Trong lý thuyết đồ thị, đồ thị có các đỉnh (hoặc nút) được nối với nhau bằng các cạnh (hoặc đường). Đối với dự án của chúng tôi, các nút là các cá nhân có liên quan và các cạnh là các kết nối của chúng. Ví dụ, nếu hai nút được kết nối với một cạnh thì có nghĩa là chúng là "bạn bè" hoặc có một số hình thức liên hệ với nhau. Sự tiếp xúc này là một con đường để bệnh lây lan. Đây là lý do tại sao chúng tôi sử dụng lý thuyết đồ thị để mô hình hóa khái niệm của mình bởi vì chúng tôi muốn xem cách dịch bệnh lây lan giữa các cá thể được kết nối trong một quần thể.
Dự án của chúng tôi cũng liên quan đến Phương pháp Monte Carlo. Phương pháp Monte Carlo là các thuật toán tạo ra việc lấy mẫu ngẫu nhiên lặp lại để nhận các kết quả số. Trong dự án của chúng tôi, chúng tôi sử dụng phương pháp này để chạy mô phỏng của chúng tôi nhiều lần thay đổi phần trăm số người chưa được tiêm chủng ban đầu để xem tỷ lệ mọi người bị nhiễm bệnh.
Tất cả mã dự án được liên kết ở dưới cùng!
Tín dụng PC:
Liên kết Matlab đến Lý thuyết Đồ thị:
Bước 1: Tạo Ma trận cận kề
Tạo một tập lệnh mới. Chúng tôi sẽ gọi là 'nhiễm trùngSim.m' của chúng tôi.
Chúng tôi sẽ tạo một biến 'NUMOFPosystem'. Bạn có thể gán nó cho bất kỳ giá trị số nguyên nào. Điều này sẽ đại diện cho số người trong dân số của bạn.
Từ bây giờ, chúng tôi sẽ giả định rằng
NUMOFPosystem = 20;
Trước tiên, hãy bắt đầu bằng cách sử dụng các hàm lý thuyết đồ thị của Matlab cho một đồ thị vô hướng.
Nếu bạn quan tâm đến việc tìm hiểu thêm, đây là một liên kết để bạn đọc chuyên sâu hơn về nó.
www.mathworks.com/help/matlab/math/diited-and-undicting-graphs.html
Đã tạo ma trận kề.
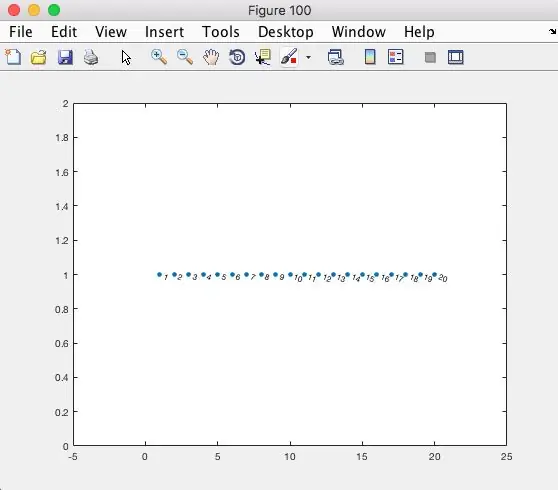
adjMatrix = số không (NUMOFPosystem);
Điều này sẽ tạo ra một ma trận vuông gồm các số 0. Mỗi hàng trong ma trận là một người. Mỗi cột trong ma trận là một người hoặc bạn bè mà người đó gặp trong ngày.
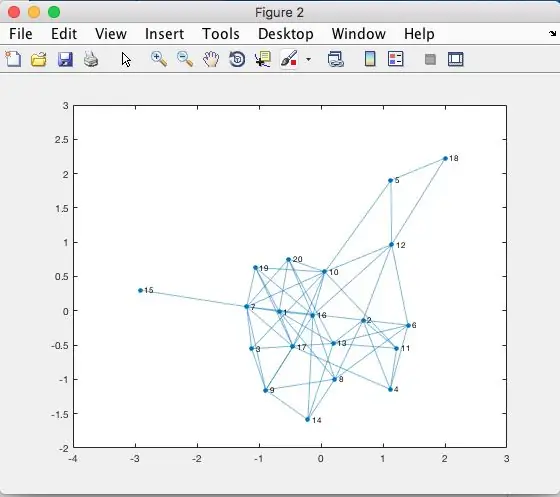
Xem Hình 100 (ở trên) để giúp hình dung adjMatrix trông như thế nào cho 20 người.
** Từ thời điểm này trở đi, chúng tôi sẽ giả sử NUMOFPosystem bằng 20. **
Bạn có thể thử vẽ đồ thị ma trận kề này. Dưới đây là một chút thông tin về cách vẽ các loại ma trận này.
Lưu ý: Cách hoạt động của ma trận kề.
Bán tại:
% tạo ma trận liền kề
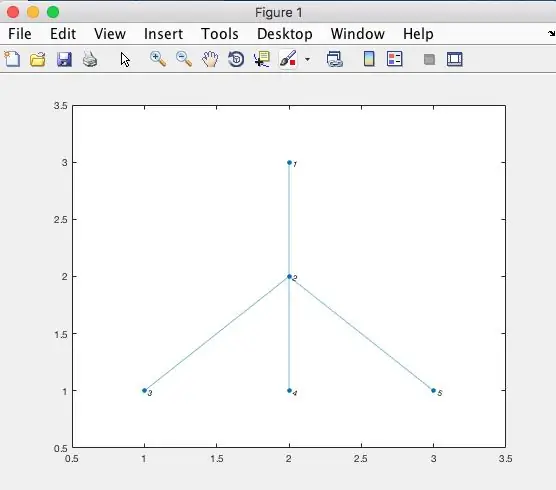
a = [0, 1, 0, 0, 0; 1, 0, 1, 1, 1; 0, 1, 0, 0, 0; 0, 1, 0, 0, 0; 0, 1, 0, 0, 0]% vẽ biểu đồ g = graph (a); % sử dụng hàm đồ thị (lý thuyết đồ thị) hình (1); h = plot (g);
Xem Hình 1 (ở trên) để biết cách thêm các cạnh trong ma trận kề, bằng cách sử dụng mã trong "Lưu ý".
Bước 2: Tạo mối quan hệ
Bây giờ những người (đỉnh hoặc nút) đã được tạo ra, chúng ta cần tạo ra một mạng lưới các quan hệ (đường hoặc cạnh của đồ thị). Điều này sẽ mô phỏng cách mọi người tương tác và gặp gỡ những người khác trong suốt một ngày.
Điều này có thể được thực hiện theo nhiều cách. Một cách để hoàn thành nhiệm vụ này trước tiên là gán một số ngẫu nhiên cho mỗi người để xác định xem mỗi người sẽ tương tác với bao nhiêu người trong một ngày.
numOfFriendsMatrix = randi ([ít bạn bèPersonCanHave, hầu hết bạn bèPersonCanHave], 1, NUMOFPooter);
Điều này tạo thành một ma trận 1 x 20 các số nguyên ngẫu nhiên đại diện cho số lượng tương tác của mỗi người trong một ngày. Các cột của ma trận này sẽ là số tương ứng với mỗi người. Ví dụ: nếu chúng ta gán ít nhất FriendsPersonCanHave = 2 và mostFriendsPersonCanHave = 5, chúng ta sẽ nhận được các giá trị ngẫu nhiên từ 2 đến 5.
Bạn gặp sự cố với randi ()? Trong thiết bị đầu cuối, nhập
giúp randi
Tiếp theo, chúng tôi tạo một ma trận ngẫu nhiên (được gọi là "allFriendsmatrix") về cách mỗi người trong quần thể được kết nối / tương tác trong quần thể.
tempMatrix = ;
đếm = 0; allFriendsMatrix = ; for k = 1: NUMOFPosystem while length (tempMatrix) ~ = numOfFriendsMatrix (k) count = count +1; temp = randi ([1, NUMOFPosystem]); tempMatrix (đếm) = tạm thời; kết thúc xóa từng độ dài while (tempMatrix) ~ = 9 tempMatrix = [tempMatrix, NaN]; end allFriendsMatrix = [allFriendsMatrix; tempMatrix]; tempMatrix = ; đếm = 0; kết thúc
Giải thích sâu về mã:
Đầu tiên chúng ta tạo một ma trận tạm thời trống để chứa danh sách bạn bè / tương tác của mỗi người. Chúng tôi cũng khởi tạo số đếm, chỉ theo dõi vị trí gắn kết nối ngẫu nhiên mới trong tempMatrix. Vòng lặp for chạy 20 lần để điều này xảy ra đối với từng người trong dân số. Vòng lặp while đầu tiên chạy cho đến khi tempMatrix của mỗi người có cùng độ dài với số lượng tương tác được chỉ định ngẫu nhiên. Trong vòng lặp này, một số ngẫu nhiên tương ứng với người trong dân số được tạo và đặt vào tempMatrix. Bởi vì độ dài của mỗi tempMatrix là khác nhau, chúng tôi cần tạo một số giá trị NaN để có thể nối tất cả các tempMatrix này thành một ma trận ('allFriendsMatrix'). Vòng lặp while thứ hai giải quyết vấn đề này bằng cách thêm NaN vào mỗi tempMatrix. Vòng lặp while được thiết lập để chạy 9 lần vì nó là một số lớn hơn 5, là giới hạn trên của bạn bè mà một người có thể được chỉ định. Giá trị '9' có thể thay đổi và có thể / phải thay đổi khi 'mostFriendsPersonCanHave' lớn hơn 9. Ba dòng mã cuối cùng (không bao gồm phần cuối) thêm tempMatrix vào hàng tiếp theo của 'allFriendsMatrix'. Sau đó, nó xóa tempMatrix và tính cho người tiếp theo.
Đầu ra
Đây là kết quả đầu ra sẽ trông như thế nào đối với lần chạy đầu tiên qua vòng lặp for (trước ba dòng cuối cùng).
tempMatrix = 16 8 17 16 13 NaN NaN NaN NaN
allFriendsMatrix =
16 8 17 16 13 NaN NaN NaN NaN 8 8 2 7 11 NaN NaN NaN NaN 10 13 NaN NaN NaN NaN NaN NaN 11 17 2 NaN NaN NaN NaN NaN 10 12 NaN NaN NaN NaN NaN NaN 4 13 2 12 NaN NaN NaN NaN NaN 17 10 9 3 1 NaN NaN NaN NaN 16 16 6 NaN NaN NaN NaN NaN NaN 3 8 17 17 14 NaN NaN NaN NaN 20 19 3 NaN NaN NaN NaN NaN NaN 13 10 NaN NaN NaN NaN NaN NaN NaN 2 18 10 16 NaN NaN NaN NaN NaN 2 6 14 3 13 NaN NaN NaN NaN 8 16 14 8 NaN NaN NaN NaN 7 7 NaN NaN NaN NaN NaN NaN 19 10 9 NaN NaN NaN NaN NaN 10 19 NaN NaN NaN NaN NaN NaN NaN 5 18 NaN NaN NaN NaN NaN NaN 1 7 NaN NaN NaN NaN NaN NaN NaN 16 7 13 10 1 NaN NaN NaN
Tiếp theo, thêm các mối quan hệ này vào adjMatrix.
cho mỗiRow = 1: NUMOFPosystem
for eachCol = 1: 9 if isnan (allFriendsMatrix (eachRow, eachCol)) == 0 adjMatrix (eachRow, allFriendsMatrix (eachRow, eachCol)) = 1; adjMatrix (allFriendsMatrix (eachRow, eachCol), eachRow) = 1; kết thúc kết thúc
Giải thích mã
Vòng lặp for kép này đi qua từng hàng và cột của 'allFriendsMatrix'. Câu lệnh if sẽ chạy cho tất cả các giá trị không phải là 'NaN'. Về cơ bản nó sẽ tạo ra các cạnh hoặc đường của đồ thị. Vì vậy, dòng đầu tiên điều này sẽ làm cho người 1 đến người 16 và người 16 thành người 1. Vì nó là vô hướng, 1 phải được thay đổi cho cả hai! Chúng ta không thể chỉ có cạnh từ 1 đến 16 mà không phải là 16 đến 1. Chúng phải đối xứng để nó chạy đúng trong Matlab.
Trong mô phỏng của chúng tôi, chúng tôi đã thiết lập rằng mọi người không thể tương tác với chính họ. Khi chúng tôi ngẫu nhiên hóa các giá trị, có khả năng ma trận liền kề của chúng tôi có lỗi này.
Hãy sửa lỗi này bằng đoạn mã sau:
cho mỗi = 1: NUMOFPosystem
adjMatrix (mỗi, mỗi) = 0; kết thúc
Giải thích mã
Vòng lặp for này đảm bảo rằng người 1 không được kết nối với người 1, người 2 không được kết nối với người 2, v.v. bằng cách tạo tất cả chúng bằng 0. Như bạn có thể thấy bên dưới trong phần đầu ra, chúng ta có đường chéo của hình vuông ma trận từ trên cùng bên trái đến dưới cùng bên phải đều là số 0.
Đầu ra
Đây là adjMatrix cuối cùng cho mô phỏng hiện tại này. Điều này chiếm tất cả các đường trong biểu đồ (Hình 2).
adjMatrix =
0 0 0 0 0 0 1 1 0 0 0 0 1 0 0 1 1 0 1 1 0 0 0 1 0 1 1 1 0 0 1 1 1 0 0 0 0 0 0 0 0 0 0 0 0 0 1 0 1 1 0 0 1 0 0 0 0 0 0 0 0 1 0 0 0 1 0 0 0 0 1 0 0 0 0 0 1 0 0 0 0 0 0 0 0 0 0 0 0 1 0 1 0 0 0 0 0 1 0 0 0 1 0 1 0 0 0 1 0 0 0 1 1 0 0 0 0 0 0 0 1 1 1 0 0 0 0 0 1 1 0 0 0 0 1 0 1 0 1 1 1 1 0 0 0 1 0 0 1 0 0 0 0 1 0 1 0 0 0 0 0 0 1 0 0 0 1 1 0 0 0 0 0 1 0 1 1 0 0 0 0 0 1 0 1 0 1 0 0 0 1 1 0 0 0 1 1 0 1 1 0 1 0 1 0 0 0 0 0 1 0 0 1 0 0 0 0 0 0 0 0 1 0 0 1 1 0 0 0 1 0 0 0 0 0 1 0 1 0 0 1 1 1 0 0 1 0 0 0 0 1 0 0 1 0 0 0 0 0 1 0 0 0 0 0 0 0 1 1 0 0 0 1 0 0 1 0 0 0 0 0 0 0 0 0 0 1 0 0 0 0 0 0 0 0 0 0 0 0 0 1 0 0 0 0 0 0 1 1 1 0 1 0 1 0 0 0 0 1 1 1 0 0 1 0 0 1 0 1 1 0 0 0 0 0 0 0 0 1 0 0 0 0 0 1 0 0 0 0 0 0 1 0 0 0 0 0 0 0 0 1 0 0 0 0 0 1 0 0 1 0 0 0 0 0 1 1 0 0 0 1 0 0 0 0 0 1 0 0 1 0 0 1 0 0 1 0 0 0 0
Xem Hình 2 để biết đồ thị của 'adjMatrix'.
Bước 3: Thêm Thống kê Bệnh tật
Giờ đây, chương trình của bạn có thể tạo một biểu đồ với một nhóm người ngẫu nhiên và tạo các mối quan hệ ngẫu nhiên, chúng tôi cần nhập thông tin hoặc số liệu thống kê của bệnh để xem những tương tác này trong một quần thể có thể làm tăng hoặc giảm lây nhiễm như thế nào.
Tạo các biến sau:
unvacc% type: double; phần trăm khả năng những người không được tiêm chủng không bị bệnh
chân không% loại: đôi; phần trăm cơ hội những người được tiêm chủng không mắc bệnh loại unvacc_perc%: double; phần trăm dân số chưa được tiêm chủng init_infect% type: int; phần trăm dân số được tiêm chủng
Tiếp theo chúng ta cần thực hiện một số phép tính.
Chúng tôi sẽ tạo một 'Nhiễm trùng', là ma trận 3 * NUMOFPosystem.
vac_perc = 1-unvacc_perc;
nhiễm trùngMat = nan (3, NUMOFPosystem); number = round (vac_perc * NUMOFPosystem); nhiễm trùngMat (1, 1: number) = tiêm chủng; nhiễm trùngMat (1, number + 1: end) = unvacc; nhiễm trùngMat (2, 1: end) = 0; nhiễm trùngMat (2, 1: init_infect) = 1;
Giải thích mã
dòng 1: Phần trăm dân số chưa được tiêm chủng được tính toán
dòng 2: tạo ma trận 3 * N số người
dòng 3: tìm hiểu số người được tiêm chủng từ tỷ lệ phần trăm được tiêm chủng
dòng 4: đối với những người được tiêm chủng, cung cấp cho họ khả năng miễn dịch liên quan đến việc có vắc xin. Giá trị này được chỉ định dựa trên nghiên cứu về bệnh.
dòng 5: đối với phần còn lại của dân số (những người chưa được tiêm chủng), hãy cung cấp cho họ phần trăm miễn dịch. Giá trị này được chỉ định dựa trên nghiên cứu về bệnh.
dòng 6: ban đầu thiết lập tất cả những người không bị nhiễm.
dòng 7: cho số người bị nhiễm ban đầu, điền vào hai cột đầu tiên cho phù hợp.
Bây giờ chúng tôi đã thiết lập tất cả các thông số cho mô phỏng bệnh, chúng tôi sẽ ngẫu nhiên hóa khả năng một người (cả tiêm chủng và chưa tiêm chủng) có bị nhiễm bệnh hay không. Điều này được thực hiện trong bước tiếp theo bằng cách gán các giá trị ngẫu nhiên từ 0 đến 1 cho mỗi người trong hàng thứ ba của 'ô nhiễm' này.
Bước 4: Chọn ngẫu nhiên Cơ hội Một Người đã Tiêm chủng và Chưa được Tiêm chủng Có thể Bị Nhiễm
Tiếp theo, gán cho mỗi người một số ngẫu nhiên, số này sẽ được sử dụng sau này để xác định xem người đó có bị nhiễm bệnh hay không.
cho w = 1: chiều dài (nhiễm trùngMat)
nhiễm trùngMat (3, w) = rand; kết thúc
Giải thích mã
Vòng lặp for này xử lý với hàng thứ ba của 'nhiễm trùng' được tạo ở bước cuối cùng. 'rand' chỉ định một giá trị từ 0 đến 1 cho mỗi chỉ mục của hàng 3.
Đầu ra
Nhiễm trùngMat hiện đã hoàn tất! Đây là với dân số được tiêm chủng 100% và có 1 người mắc bệnh ban đầu.
Nhiễm trùngMat =
Cột 1 đến cột 12 0,7500 0,7500 0,7500 0,7500 0,7500 0,7500 0,7500 0,7500 0,7500 0,7500 0,7500 0,7500 1,0000 0 0 0 0 0 0 0 0 0 0 0 0,0869 0,5489 0,3177 0,9927 0,7500 0,7500 0,7500 0,7500 0,7500 0,7500 0,7500 0,7500 0,7500 1,0000 0 0 0 0 0 0 0 0 0 0 0,0869 0,5489 0,3177 0,9927 0,7236 0,5721 0,7172 0,9766 0,4270 0,9130 0,8973 0,7500 0,7500 0,7500 0,7500 0,7500 0,7500 0 0 0 0 0 0 0 0 0,0480 0,3593 0,2958 0,6291 0,1362 0,3740 0,8648 0,2503
hàng 1: Phần trăm cơ hội KHÔNG mắc bệnh
hàng 2: Bị nhiễm hoặc không bị nhiễm (giá trị boolean)
hàng 3: Số dùng để kiểm tra xem người không bị nhiễm có bị nhiễm bệnh hay không nếu họ gặp người bị nhiễm bệnh. Nếu người chưa bị nhiễm mà gặp người bị nhiễm mà con số này lớn hơn số ở hàng 1 (đối với cùng một cột) thì họ đã bị nhiễm. Chúng tôi sẽ mã hóa chức năng này trong bước 7.
Bước 5: Tạo ma trận những người chưa được tiêm chủng và bị nhiễm bệnh từ thông tin ban đầu
Tạo 2 ma trận được gọi là "matrixUnvacc" và "matrixInfected" để lưu trữ tất cả những người bị nhiễm từ lây nhiễm. Điều này sẽ được sử dụng để chúng tôi có thể mã màu biểu đồ của những người bị nhiễm bệnh, chưa được tiêm chủng hoặc đã được tiêm chủng, giúp hình dung tác động của những người chưa được tiêm chủng so với những người đã được tiêm chủng.
xóa từng
matrixInfected = ; ma trậnUnvacc = ; for h = 1: length (deliveryMat) if NhiễmMat (1, h) == unvacc matrixUnvacc = [matrixUnvacc, h]; end end cho person = 1: NUMOFPosystem if conferenceMat (2, person) == 1 matrixInfected = [matrixInfected, person]; kết thúc cuối cùng
Giải thích mã
Tạo hai ma trận trống để lưu trữ số người chưa được chủng ngừa và số người bị nhiễm bệnh, tương ứng. Cả hai vòng lặp for đều chạy 20 lần và nếu câu lệnh if được thỏa mãn, thì số đó sẽ được thêm vào ma trận chính xác.
Đầu ra
matrixUnvacc =
matrixInfected =
[1]
Bước 6: Vẽ đồ thị ban đầu
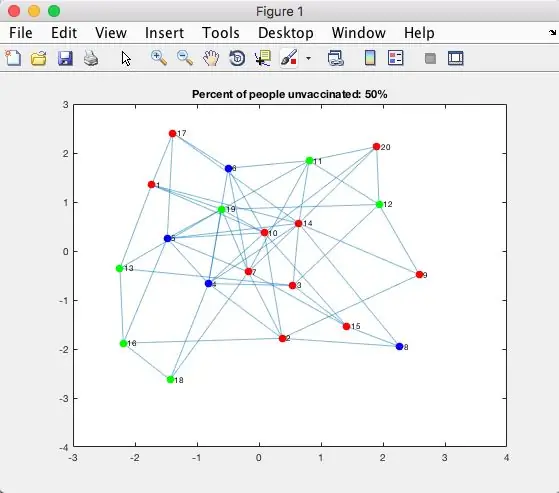
Tiếp theo, chúng ta sẽ vẽ ma trận kề.
g = graph (adjMatrix);
hình (1) p = plot (g, 'NodeColor', 'b', 'MarkerSize', 7); highlight (p, matrixUnvacc, 'NodeColor', 'g') highlight (p, matrixInfected, 'NodeColor', 'r') title_unvacc = unvacc_perc * 100; title (['Phần trăm số người chưa được tiêm:', num2str (title_unvacc), '%']); tạm dừng (tốc độ)
Giải thích mã
Lý thuyết đồ thị trong Matlab đã được tích hợp sẵn các hàm. Khi chúng tôi sử dụng hàm graph (), chúng tôi có thể dịch 'adjMatrix' thành một biểu đồ vô hướng thực tế. Sau đó, chúng ta phải tạo một âm mưu bằng cách sử dụng hàm plot () để thực sự xem nó trông như thế nào. Chúng tôi đặt biểu đồ này () thành một biến để chúng ta có thể thao tác và thay đổi màu sắc của âm mưu dễ dàng hơn trong suốt quá trình mô phỏng. Tất cả mọi người (hoặc các nút) ban đầu được đặt thành màu 'xanh lam'. Tiếp theo, tất cả những người chưa được tiêm chủng được đặt thành màu 'xanh lá cây'. Những người bị nhiễm bệnh sau đó được đặt thành màu 'đỏ'. Tiêu đề được đặt theo giá trị phần trăm nhất định của những người chưa được tiêm chủng đang được kiểm tra. Hàm pause () tạm thời dừng việc thực thi MatLab. Chúng tôi chuyển qua tốc độ thay đổi được lan truyền được tính bằng giây.
Xem hình (ở trên) để xem biểu đồ được mã hóa màu ngẫu nhiên.
Tìm hiểu thêm về hàm highlight () trong MatLab.
Bước 7: Mô phỏng sự tiến triển của nhiễm trùng
Tiếp theo, chúng ta cần tìm ra ai bị nhiễm sau các tương tác (được ghi lại trong adjMatrix) và cập nhật biểu đồ khi ai đó bị nhiễm.
Sử dụng adjMatrix để xác định những người nào bị nhiễm sau khi họ tương tác với mọi người trong một ngày.
for eachRow = 1: length (adjMatrix)
if nhiễmMat (2, eachRow) == 1 cho mỗiCol = 1: length (adjMatrix) nếu adjMatrix (mỗiRow, mỗiCol) == 1% mỗiRow = người% mỗiCol = bạn của nó% bạn của mỗi người và xem họ có bị nhiễm bệnh hay không. nếu nhiễm trùngMat (3, mỗiCol)> nhiễmMat (1, mỗiCol) nhiễmMat (2, mỗiCol) = 1; tô sáng (p, mỗiCol, 'NodeColor', 'r') tạm dừng (tốc độ) kết thúc kết thúc kết thúc kết thúc
Vòng lặp for lặp lại qua từng người. Nó kiểm tra xem người đó có bị nhiễm bệnh hay không, nó sẽ kiểm tra từng người / bạn bè mà họ đã tương tác và kiểm tra xem mức độ miễn dịch của người bạn đó có lớn hơn mức độ mắc bệnh hay không. Đây là nơi phát huy tác dụng của 'Nhiễm trùngMat' mà chúng tôi đã tạo trước đó. Hàng thứ nhất và thứ 3 của mỗi cột của người bạn được so sánh và nếu hàng thứ 3 lớn hơn, điều đó có nghĩa là người bạn đó không có khả năng miễn dịch đủ cao để thoát khỏi bệnh tật và cuối cùng bị nhiễm bệnh. Chúng tôi cũng thay đổi màu sắc bằng cách sử dụng highlight () thành màu đỏ nếu chúng bị nhiễm trùng.
Bây giờ mã của bạn cho mô phỏng sẽ hoạt động! và đối với bất kỳ quy mô dân số nào, chỉ cần thay đổi NUMOFPosystem!
Bước 8: Sử dụng lý thuyết Monte Carlo
Để tiến thêm một bước nữa và trích xuất dữ liệu từ trình mô phỏng của chúng tôi ('nhiễm trùngSim.m'), chúng tôi muốn tính toán và vẽ biểu đồ xu hướng phần trăm số người chưa tiêm phòng bị nhiễm bệnh và phần trăm số người được tiêm phòng bị nhiễm bệnh. Chúng tôi đưa ra giả thuyết rằng phần trăm những người được tiêm phòng bị nhiễm bệnh sẽ thấp hơn rất nhiều so với phần trăm của những người không được tiêm phòng bị nhiễm bệnh.
Bước 9: Tạo tệp ('Nhiễm trùngSim.m') với Mô phỏng thành một chức năng
Để chạy Monte Carlo, chúng tôi muốn chạy mô phỏng nhiều lần và tích lũy dữ liệu để có thể sử dụng dữ liệu đó để vẽ biểu đồ phần trăm số người bị nhiễm bệnh.
Hàm có thể được thiết lập như sau:
chức năng đầu ra = lây nhiễmSim (unvacc, vac, NUMOFPosystem, unvacc_perc, init_infect, speed)
Nhận xét các biến trong mô phỏng của bạn vì bây giờ bạn đang chuyển các biến này qua tệp chính (chúng tôi sẽ bắt đầu viết điều này trong bước 12):
unvacc, vacxin, NUMOFPosystem, unvacc_perc, init_infect
Biến mới
tốc độ, vận tốc
sẽ được gán trong tệp chính (Monte_Carlo.m).
Lưu ý: Đừng quên phần cuối ở cuối tệp chức năng để kết thúc chức năng!
Bước 10: Tính Tỷ lệ người chưa được tiêm chủng và người chưa được tiêm chủng bị nhiễm bệnh
Điều này tính toán tỷ lệ phần trăm những người không được tiêm chủng đã bị nhiễm bệnh. Mã này nằm ở cuối tệp 'nhiễm trùngSim.m'.
number_of_unvacc = 0;
number_of_infec_unvacc = 0; % tính toán tỷ lệ phần trăm những người chưa được tiêm chủng đã bị nhiễm bệnh cho x = 1: length (Nhiễm trùngMat) nếu Nhiễm trùngMat (1, x) == unvacc number_of_unvacc = number_of_unvacc + 1; kết thúc nếu nhiễm trùngMat (1, x) == unvacc & nhiễm trùngMat (2, x) == 1 number_of_infec_unvacc = number_of_infec_unvacc +1; end end %_of_unvacc_and_infec = (number_of_infec_unvacc / number_of_unvacc) * 100;
Giải thích mã
Trong vòng lặp for, nó sẽ lặp lại NUMOFPranty lần. Mỗi lần con số trong nhiễm trùngMat tương ứng với số unvacc (tức là 0,95 == 0,95), thì số người chưa được tiêm chủng sẽ tăng lên 1. Mỗi lần con số trong bệnh nhiễmMat tương ứng với số unvacc và họ bị nhiễm, số người mắc và chưa tiêm phòng tăng 1. Dòng cuối cùng chia số người mắc, chưa tiêm cho tổng số người chưa tiêm. Sau đó, tỷ lệ phần trăm được tính từ này.
Thử thách:
Hãy thử tính toán phần trăm số người đã được tiêm phòng! (Gợi ý: nó rất giống với đoạn mã trên, tuy nhiên một số biến được thay đổi và tên được điều chỉnh.)
Tiếp theo, phần trăm số người bị nhiễm bệnh dựa trên tổng dân số được tính:
pre_per_infect = cumsum (Nhiễm trùngMat (2,:));
per_infect = (pre_per_infect (1, NUMOFPosystem) / NUMOFPosystem) * 100;
Giải thích mã
Tổng tích lũy được tính bằng cách sử dụng hàng thứ hai của ô nhiễm, lưu trữ các giá trị 1 và 0 tùy thuộc vào việc người đó có bị nhiễm bệnh hay không. Vì hàm cumsum () trả về một ma trận, chúng tôi lấy giá trị cuối cùng trong ma trận ('pre_per_infect (1, NUMOFPosystem)'), giá trị này phải là tổng thực tế của tất cả các giá trị từ 'Nhiễm trùng (2,:)' '. Bằng cách chia tổng cho NUMOFPosystem và nhân nó với 100, chúng tôi nhận được phần trăm cuối cùng của người bị nhiễm trong tổng số dân.
Bước 11: Tạo một biến đầu ra trong hàm 'Nhiễm trùngSim.m' của bạn
đầu ra = [per_infect,erc_of_unvacc_and_infec, phần trăm_of_vacc_and_infec];
Giải thích mã
Lưu trữ thông tin này trong đầu ra, thông tin này sẽ được gửi lại vào main (Monte_Carlo.m) khi hàm được gọi và chạy xong. Dữ liệu này được sử dụng để vẽ biểu đồ phần trăm số người bị nhiễm bệnh của những người đã được tiêm chủng và chưa được tiêm chủng.
Chức năng 'Nhiễm trùngSim.m' của bạn sẽ được thực hiện ngay bây giờ! Tuy nhiên, nó sẽ không chạy vì chúng ta vẫn cần viết main!
Bước 12: Tạo Menu để Lấy Điều kiện ban đầu của Mô phỏng từ Người dùng
Hãy nhớ cách chúng ta đã nói về biến
tốc độ, vận tốc
sẽ được tạo và chuyển qua hàm chính? Chúng ta cần lấy các giá trị để truyền vào hàm. Lưu ý, thứ tự của các giá trị khi gọi hàm có ý nghĩa!
Bắt đầu bằng cách yêu cầu người dùng nhập một số câu trả lời vào thiết bị đầu cuối.
> Chọn bệnh. Lưu ý rằng nó có phân biệt chữ hoa chữ thường >> Ho gà >> Cúm >> Sởi >> Bệnh được chọn: Cúm >> Chọn quy mô dân số. >> 20 >> 200 >> Dân số được chọn: 20 >> Chọn tốc độ mô phỏng. >> Nhanh >> Chậm >> Tốc độ được chọn: Nhanh
Đoạn mã dưới đây hỏi người dùng họ muốn xem xét bệnh gì.
disp ('Chọn một bệnh. Lưu ý rằng nó phân biệt chữ hoa chữ thường')
fprintf ('Pertussis / nFlu / nMeasles / n') disease = input ('Bệnh được chọn:', 's'); if isequal (bệnh, 'Ho gà') vacxin =.85; % 15% khả năng mắc bệnh unvacc =.20; % 80% khả năng mắc bệnh khác nếu tiêm vắc xin là hậu quả (bệnh, 'Cúm') = 0,75; % 25% khả năng mắc bệnh unvacc =.31; % 69% khả năng mắc bệnh khác nếu tiêm vắc xin là hậu quả (bệnh, 'Sởi') =.97; % 3% khả năng mắc bệnh unvacc =.10; % 90% khả năng hết bệnh
Giải thích mã:
Hàm disp () in câu lệnh ra màn hình và nó cũng in ra các tùy chọn khác nhau. Bệnh sẽ được chỉ định cho phù hợp. Phiên bản này hiện không giải thích cho đầu vào không hợp lệ. Đầu vào không hợp lệ sẽ tạo ra lỗi và dừng chương trình hoàn toàn. Mỗi bệnh đều có các giá trị vắc xin và không tiêm vắc xin đi kèm. Các giá trị này KHÔNG phải là ngẫu nhiên. Chúng tôi có được những giá trị này từ việc nghiên cứu số liệu thống kê về các căn bệnh.
Tiếp theo, chúng tôi cần hỏi người dùng xem họ muốn kiểm tra quy mô dân số lớn hay nhỏ cho căn bệnh họ đã chọn.
disp ('Chọn kích thước dân số.')
fprintf ('20 / n200 / n ') speed = input (' Dân số được chọn: ',' s '); if isequal (speed, '20') civil_size = 20; elseif isequal (tốc độ, '200') dân số_size = 200; kết thúc
Giải thích mã
Điều này sẽ in ra một tuyên bố cho người dùng và yêu cầu người dùng nhập kích thước dân số mà họ muốn kiểm tra. Phiên bản này hiện không giải thích cho đầu vào không hợp lệ. Đầu vào không hợp lệ sẽ tạo ra lỗi và dừng chương trình hoàn toàn. 20 người được chọn vì đây là kích thước mẫu nhỏ nên vẫn có ý tưởng tốt về cách lây nhiễm trong một quần thể nhỏ. 200 người được chọn là lựa chọn lớn hơn vì 200 điểm được vẽ trên biểu đồ hầu như không có bất kỳ điểm trùng lặp nào để mọi thứ có thể dễ dàng được nhìn thấy và phân biệt với nhau.
Tiếp theo, chúng ta cần tìm tốc độ của mô phỏng.
disp ('Chọn tốc độ mô phỏng.')
fprintf ('Fast / nSlow / n') speed = input ('Tốc độ đã chọn:', 's'); if isequal (speed, 'Fast') sim_speed = 0; elseif isequal (tốc độ, 'Chậm') sim_speed = 0,25; kết thúc
Giải thích mã
Quá trình này cũng giống như việc xác định loại bệnh và quy mô dân số. Đối với nhanh chóng, sẽ không có tạm dừng. và đối với chậm, sẽ có độ trễ 0,25 giây trong vòng lặp for khi chạy mô phỏng.
Tuyệt vời! Bây giờ chúng tôi có tất cả các đầu vào từ người dùng mà chúng tôi cần! Hãy chuyển sang thu thập dữ liệu về các tỷ lệ phần trăm khác nhau của những người chưa được tiêm chủng.
Bước 13: Chọn% số người chưa được tiêm chủng và tính toán tỷ lệ trung bình của số người chưa được tiêm chủng và bị nhiễm bệnh cho phần trăm được chọn
Mã này dành cho 0% những người chưa được tiêm chủng.
% -------% 0 Chưa tiêm phòng ------------
per_infect_av_0 = ; %_of_unvacc_and_infec_av_0 = ; cho i = 1:20 out = Nhiễm trùngSim (unvacc, vắc xin, kích thước dân số, 0, 1, sim_speed); per_infect_av_0 = [per_infect_av_0, out (1, 1)]; %_of_unvacc_and_infec_av_0 = [%_of_unvacc_and_infec_av_0, hết (1, 2)]; end average_infected_0 = mean (per_infected_av_0); Average_unvacc_and_infected_0 = mean (phần trăm_của_unvacc_and_infec_av_0);
Giải thích mã:
Vòng lặp for được chạy 20 lần. Đầu ra từ hàm, Nhiễm trùngSim (), được lưu trữ ở ngoài. Mỗi khi vòng lặp for chạy, phần trăm bị nhiễm bệnh trong tổng dân số sẽ được thêm vào ma trận, 'per_infect_av_0'. Ngoài ra, tỷ lệ phần trăm chưa được tiêm chủng và bị nhiễm bệnh cũng được thêm vào mỗi lần vào ma trận 'phần trăm_of_unvacc_and_infec_av_0'. Trong hai dòng cuối cùng, hai ma trận nói trên sau đó được tính trung bình và lưu trữ trong các biến. Tóm lại, tỷ lệ phần trăm được lưu trữ cho mỗi mô phỏng, tính trung bình và vẽ biểu đồ. Monte Carlo được sử dụng để hiển thị giá trị trung bình của việc chạy mô phỏng và hiển thị kết quả. Đối với mục đích thử nghiệm của chúng tôi, chúng tôi chọn chạy mô phỏng 20 lần và tính trung bình các giá trị đó.
Thử thách:
Lặp lại cho tất cả các tỷ lệ phần trăm bạn muốn kiểm tra! Điều này có thể được thực hiện bằng cách thay đổi tên biến theo số phần trăm. Chúng tôi đã kiểm tra 0%, 5%, 10%, 20%, 30% và 50%.
Gợi ý:
Dòng duy nhất phải được thay đổi trong mã thực là
out = Nhiễm trùngSim (unvacc, vắc xin, kích thước dân số, 0, 1, sim_speed);
Thay đổi số 0 thành phần trăm ở dạng thập phân. Ví dụ: đối với mô phỏng 5% không được tiêm chủng, số 0 nên được thay thế bằng 0,5.
Bước 14: Biểu đồ: 'Xu hướng lây nhiễm ở những bệnh nhân chưa được tiêm chủng Vs. Được chủng ngừa cho các bệnh được chỉ định '
Đây là mã để tạo biểu đồ về xu hướng lây nhiễm ở những người chưa được tiêm chủng so với những người chưa được tiêm chủng.
graph_mat_y = [Average_infected_0, Average_infected_5, Average_infected_10, Average_infected_20, Average_infected_30, Average_infected_50];
graph_mat_x = [0, 5, 10, 20, 30, 50]; độ dốc = (trung bình_độ_độ_5-trung_bình_tính_0) / 5; line_y = [average_infected_0, (angle * 50) + average_infected_0]; line_x = [0, 50]; hình (2) plot (graph_mat_x, graph_mat_y); line (line_x, line_y, 'Color', 'red', 'LineStyle', '-'); title (['Xu hướng không tiêm chủng cho', bệnh tật]); xlabel ('Tỷ lệ phần trăm chưa được tiêm chủng ban đầu'); ylabel ('Phần trăm nhiễm trùng cuối cùng')
Giải thích mã
dòng 1: các giá trị y được gán cho mức trung bình của phần trăm bị nhiễm
dòng 2: các giá trị x được chỉ định cho tỷ lệ phần trăm ban đầu chưa được tiêm chủng
dòng 3: tính độ dốc 0% và 5%
dòng 4: lưu trữ các giá trị y của dòng. Đây là phần tiếp theo của phần 0% đến 5%.
dòng 5: lưu trữ các giá trị y của dòng. Đường này kéo dài theo chiều dài của biểu đồ.
dòng 6: tạo hình
dòng 7: vẽ biểu đồ giá trị x và y của phần trăm bị nhiễm, chưa được tiêm phòng.
dòng 8: vẽ đường thẳng. Điều này được sử dụng để chỉ ra rằng nó không tăng theo tuyến tính mà theo cấp số nhân.
dòng 9: Đặt tiêu đề cho biểu đồ.
dòng 10-11: Đặt nhãn x và y cho đồ thị.
Bây giờ bạn sẽ có thể thấy rằng tỷ lệ dân số không được tiêm chủng càng lớn thì số lượng nhiễm trùng càng lớn. Bạn cũng sẽ thấy rằng hầu hết các chấm chuyển sang màu đỏ là các chấm màu xanh lá cây, cho thấy rằng vắc-xin có ích ở một mức độ nào đó! Hy vọng bạn thích hướng dẫn này. Bình luận nếu bạn có bất kỳ câu hỏi nào!
Bước 15: Sản phẩm cuối cùng: Mô phỏng trông như thế nào
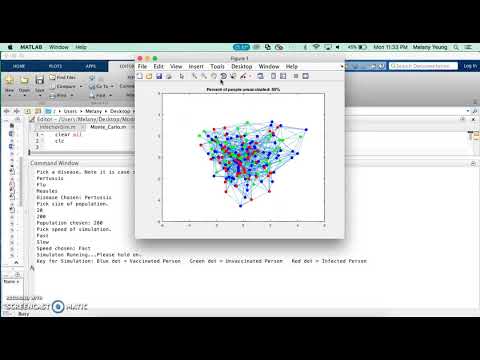
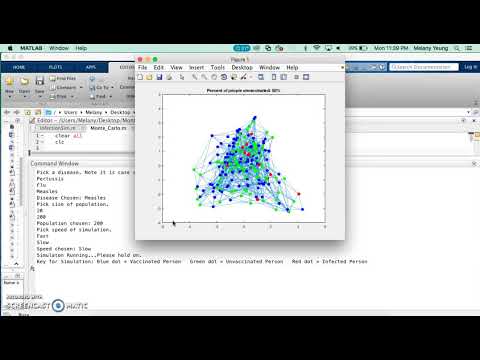
Tất cả mã có thể được tìm thấy ở đây
Đề xuất:
Máy phát Flysky RF được cung cấp năng lượng thông qua kết nối tín hiệu dây USB + với PC + Phần mềm giả lập miễn phí: 6 bước

Bộ phát RF Flysky được hỗ trợ qua kết nối tín hiệu dây USB + với PC + Phần mềm giả lập miễn phí: Nếu bạn giống tôi, bạn sẽ thích thử nghiệm bộ phát RF của mình và tìm hiểu trước khi bạn gặp sự cố máy bay / máy bay không người lái RF thân yêu của mình. Điều này sẽ mang lại cho bạn niềm vui bổ sung, đồng thời tiết kiệm rất nhiều tiền và thời gian. Để làm như vậy, cách tốt nhất là kết nối thiết bị phát RF với bạn
Phiên bản không dây của Cửa nhà để xe của tôi mở hay đóng ?: 7 bước

Phiên bản không dây của … Cửa nhà để xe của tôi đang mở hay đã đóng ?: Chúng tôi muốn có một hệ thống chỉ dẫn đơn giản, rẻ và đáng tin cậy cho chúng tôi biết cửa nhà để xe của chúng tôi đang mở hay đóng. Có rất nhiều " Cửa nhà để xe của tôi có mở không " các dự án. Phần lớn các dự án này là có dây cứng. Trong trường hợp của tôi thì
Giám sát nhiệt độ phòng có điều khiển Raspberry Pi với đầu ra hình ảnh Gnuplot và khả năng cảnh báo qua email: 7 bước
Khả năng giám sát nhiệt độ phòng có điều khiển bằng Raspberry Pi Với khả năng xuất hình ảnh và thông báo qua email của Gnuplot: Nơi tôi làm việc, có một căn phòng rất quan trọng chứa rất nhiều máy tính. Nhiệt độ môi trường xung quanh của căn phòng này phải rất mát mẻ để tối ưu hóa hiệu suất của các hệ thống này. Tôi được yêu cầu đưa ra một hệ thống giám sát có khả năng
Bỏ qua các hệ thống lọc trên máy tính mà không có sự không an toàn của TOR (Bộ định tuyến củ hành) hoặc đường hầm Internet qua SSH: 5 bước
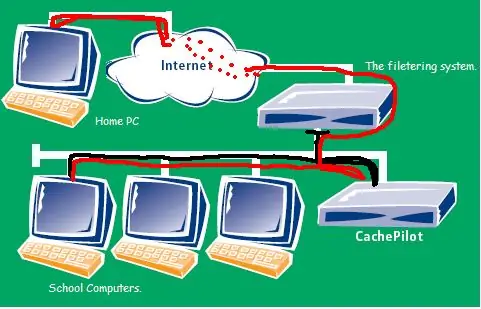
Bỏ qua Hệ thống lọc trên máy tính mà không có sự không an toàn của TOR (Bộ định tuyến củ hành) hoặc Đường hầm Internet qua SSH: Sau khi đọc một bài đăng về bộ định tuyến củ hành (tor) cho phép bạn vượt qua kiểm duyệt mà không bị theo dõi, tôi đã rất ngạc nhiên. Sau đó, tôi đọc rằng nó không an toàn lắm vì một số nút có thể nhập dữ liệu sai và trả lại các trang sai. Tôi đã nghĩ đến myse
Giá đỡ máy tính xách tay giá rẻ tự làm thông qua tủ quần áo

Giá đỡ máy tính xách tay giá rẻ tự làm qua TheClosetEntosystemur.com: CẬP NHẬT (19/11/2008): Tôi đã tạo một mẫu PDF có thể in được mà bạn có thể sử dụng để trợ giúp quá trình cắt bìa cứng. Kích thước của mẫu hơi khác so với hướng dẫn vì tôi quyết định tạo các móc phía trước giữ t
